Etude de l'action des rayonnements
ultraviolets sur Serratia marcescens
 Serratia marcescens est une bactérie non pathogène (opportuniste) qui produit
naturellement un pigment rouge sang à activité antibiotique et immunosuppressive
: la prodigiosine. La prodigiosine est produite en tant que métabolite
secondaire sous l'influence du système général d'expression des gènes appelé "quorum
sensing". La croissance en milieu liquide à une faible densité
cellulaire permet une expression de bas niveau d'un régulateur positif de
l'expression de la prodigiosine. La membrane des bactéries est perméable à ce
régulateur. La concentration cellulaire de ce régulateur qui pénètre par
diffusion reste donc faible lorsque la densité cellulaire est faible. Lorsque
la densité cellulaire augmente, la concentration intracellulaire du régulateur
augmente jusqu'à déclencher la production massive de prodigiosine. Il se
produit le même phénomène en culture sur milieu solide et la coloration
n'apparaît que pour des colonies ayant un diamètre supérieur à 1 mm.
Serratia marcescens est une bactérie non pathogène (opportuniste) qui produit
naturellement un pigment rouge sang à activité antibiotique et immunosuppressive
: la prodigiosine. La prodigiosine est produite en tant que métabolite
secondaire sous l'influence du système général d'expression des gènes appelé "quorum
sensing". La croissance en milieu liquide à une faible densité
cellulaire permet une expression de bas niveau d'un régulateur positif de
l'expression de la prodigiosine. La membrane des bactéries est perméable à ce
régulateur. La concentration cellulaire de ce régulateur qui pénètre par
diffusion reste donc faible lorsque la densité cellulaire est faible. Lorsque
la densité cellulaire augmente, la concentration intracellulaire du régulateur
augmente jusqu'à déclencher la production massive de prodigiosine. Il se
produit le même phénomène en culture sur milieu solide et la coloration
n'apparaît que pour des colonies ayant un diamètre supérieur à 1 mm.
La synthèse de la prodigiosine s'effectue en plusieurs
étapes, codées par des gènes qui peuvent chacun être affectés par les UV (mutation)
et produire une pigmentation incomplète (pigment orange) ou même l'absence de
pigmentation.
Il faut noter qu'un même phénotype
peut provenir de mutations à des niveaux différents dans la voie de synthèse
(génotypes différents).
Les objectifs de ce TP sont :
-
d'isoler des mutants pigmentaires de Serratia après
exposition à des agents mutagènes physiques, ici les UV.
-
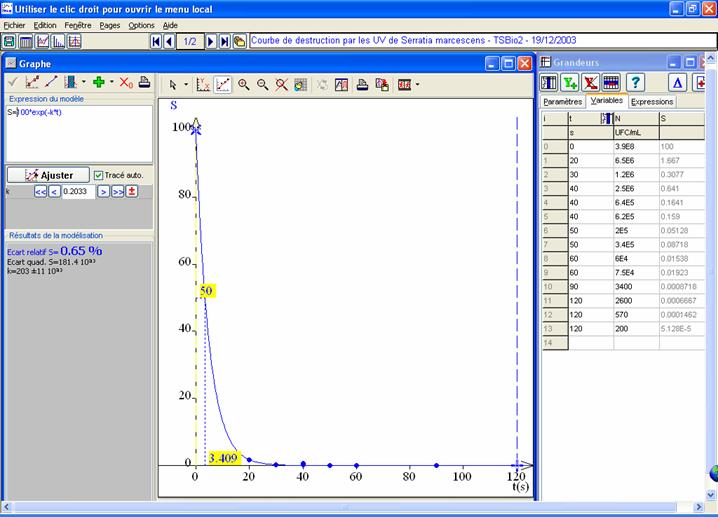
de tracer la courbe de destruction de Serratia sous
l'action des UV en fonction du temps d'exposition : log(N)=f(t).
-
d'établir le spectre d'absorption des différents pigments
produits.
-
caractériser les différents pigments par syntrophie (échanges
de nutriments ou de facteurs de régulation)
Souches et milieux :
-
100 mL de culture de 18 heures de Serratia marcescens
en BN
-
tubes de 9 ou 10 mL d'eau physiologique stériles
-
boîtes de milieu PG : peptone 5 g, glycérol 10 mL, agar 20
g, Eau qsp 1 Litre
-
Lampe UV proche de 260 nm (254 nm par exemple) et
protection individuelle
-
Spectrophotomètre et cuves en verre ;
-
acétone
JOUR 1 :
-
Centrifuger 10 minutes à 5-7000 rpm 10 mL d'une culture de
18 heures de Serratia marcescens en bouillon pour culotter les cellules,
-
Resuspendre le culot dans 10 mL d'eau physiologique
stérile et transférer dans une boîte de Pétri,
On peut aussi mettre en suspension
une oese de culture dans 9 mL d'eau physiologique.
-
Déposer la boîte couvercle ouvert sur un plateau oscillant
sous une hotte à flux laminaire (PSM2),
-
Irradier avec des UV autour de 260 nm (lampe à
254 nm, maintenue à 20 cm au dessus de la boîte) en prélevant des
échantillons à 0, 30, 60, 90 et 120 s (un temps par binôme). Porter
une protection contre les UV !
-
Réaliser des dilution décimales jusqu'à 10-5 et
étaler 0,1 mL de chaque sur 2 boîtes de milieu PG.
-
Incuber à température ambiante près d'une fenêtre en
évitant l'exposition directe (le pigment n'est pas produit à 37°C : une
température de 25-30°C est optimale, et la production de pigment est augmentée
en présence de lumière, mais attention à la photolyase !)
JOUR 2 :
-
Rechercher les mutants pigmentaires sur les boîtes et les
réisoler sur de nouvelles boîtes. Attention à ne pas confondre des contaminants
avec des mutants apigmentés ! Entourer les boîtes de parafilm.
JOUR 3 :
-
émulsionner chaque mutant dans du méthanol acidifié à pH
2.5 pour dissoudre les pigments, centrifuger et tracer son spectre d'absorption
entre 350 et 800 nm.
-
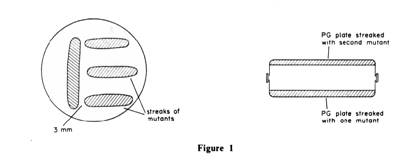 Identification des différents mutants :
Identification des différents mutants :
o réaliser des
stries des différents mutants comme indiqué sur le schéma de gauche pour
caractériser les intermédiaires solubles
o pour rechercher
les intermédiaires volatils, isoler un mutant sur une boîte de milieu PG
et faire des stries avec plusieurs autres mutants sur une autre boîte. Eliminer
les couvercles des boîtes et les maintenir ensemble avec du scotch.
o Incuber toutes les
boîtes pendant 1 à 2 jours.
Résultats :
-
tracer la courbe % survivants=f(t) et la droite
log(N)=f(t) où N = nombre UFC/mL pour chaque temps d'exposition.
-
Compter les différents mutants pigmentaires obtenus et
tracer le spectre d'absorption des différents pigments mutés.
-
Interpréter les expériences de syntrophie et conclure.
DOCUMENTS

Localisation de points de mutation dans la voie de
biosynthèse de la prodigiosine. La prodigiosine est le pigment habituel rouge
et la norprodigiosine est le pigment orange produit par les mutants de classe
B3. MAP = 2-méthyl-3-n-amyl pyrole ; MBC =
4-methoxy-2,2'-bipyrole-5-carboxyaldéhyde ; HB =
4-hydroxy-2'-bipyrole-5-carboxyaldéhyde.
|
|
|
|
Donneur |
|||||
|
|
|
couleur du mutant |
C |
B1 |
B2 |
B3 |
M1 |
M2 |
|
Accepteur |
C |
rose |
|
- |
- |
- |
- |
- |
|
B1 |
blanc ou jaune |
S |
|
S |
S |
S |
S |
|
|
B2 |
rose ou blanc |
S |
- |
|
S |
S |
S |
|
|
B3 |
orange |
S |
- |
- |
|
S |
S |
|
|
M1 |
rose pâle |
V |
V |
V |
V |
|
V |
|
|
M2 |
rose ou blanc |
V |
V |
V |
V |
- |
|
|
La pigmentation rouge de la souche
acceptrice est due :
soit à un produit volatil (V) ; soit
à un produit soluble (S) ; - : pas de pigmentation.

Absorbance spectra of bacterial cultures. The figure
is a composite of visible spectra from S. marcescens Nima, S. marcescens ampicillin-resistant Mutant 5 (non pigmented), and P.
aeruginosa grown under conditions that induce prodigiosin expression. Cells
from day-old slant cultures were grown to saturation in nutrient broth + 0.5%
maltose. They were diluted in this same medium to optical densities within the
linear range for both prodigiosin and whole cell quantification. Each sample
was scanned at a speed of 20 nm per second.
Figure 1 also
indicates that optical density values at 620 nm (OD620) can be used to measure
cell density without interference due to prodigiosine absorbance.
Quelques
résultats et valeurs de référence :